统计与大数据研究院罗翔宇老师依托校级计算服务发表高水平论文
中国人民大学统计与大数据研究院罗翔宇老师和合作者提出了一个统计分层和去卷积方法(称为HIRE)用于探测表观基因组关联分析中具有细胞类型特异性的风险CpG位点。此研究成果的计算资源由中国人民大学校级高性能与大数据公共计算云计算平台等单位提供。以往的统计方法仅仅探测出风险CpG位点而并不能知晓位点所处的细胞类型信息,且伴随着较低的灵敏度。HIRE不仅提高了风险CpG位点探测的灵敏度,更能通过其分层结构得到风险CpG位点所处的细胞类型信息。相关研究成果以“Detection of cell-type-specific risk-CpG sites in epigenome-wide association studies”在线发表在《自然·通讯》(Nature Communications)上。
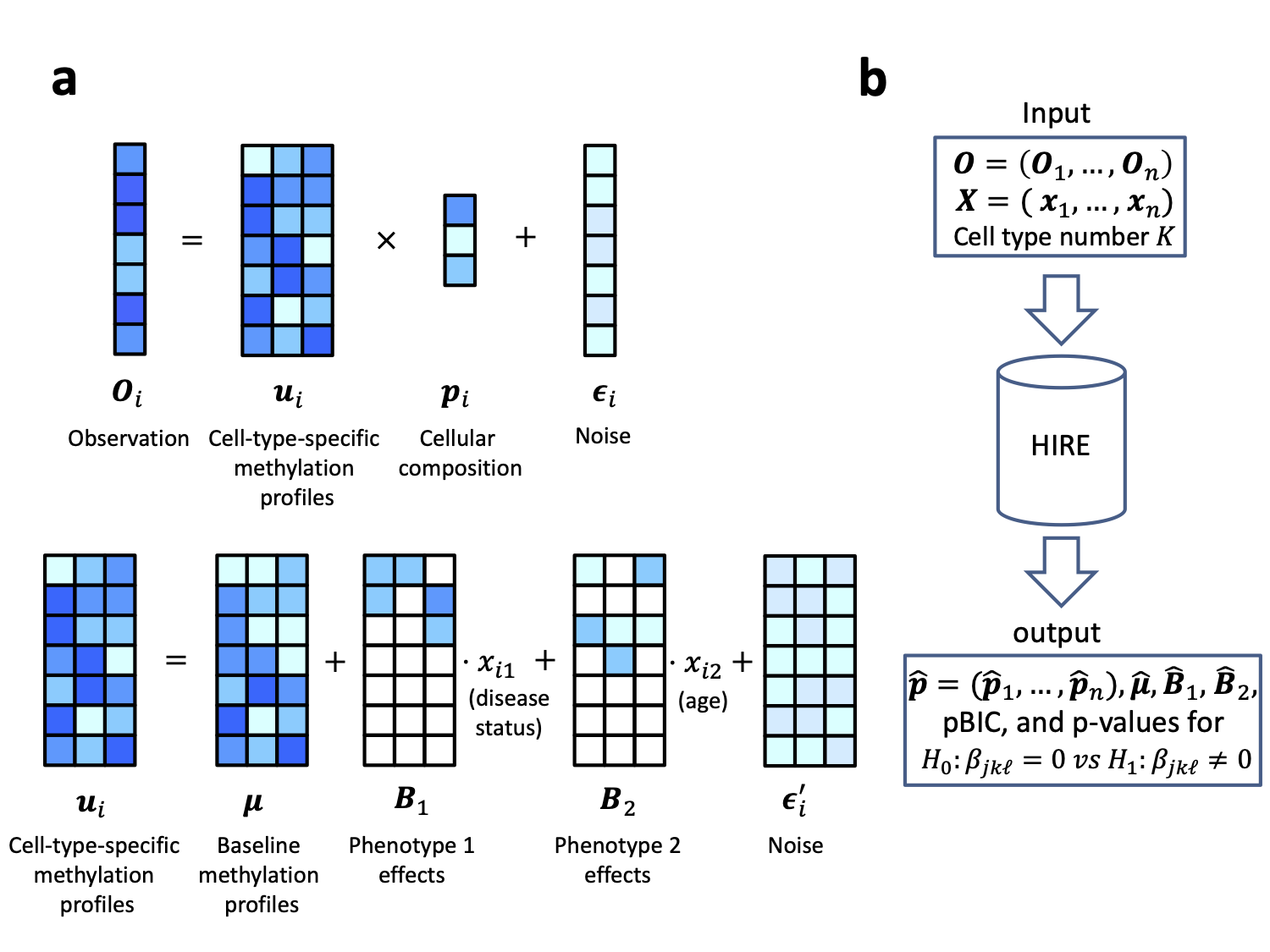 在表观基因组中,一个重要的基本概念是DNA甲基化,它是指DNA序列上CpG二核苷酸的胞嘧啶与甲基基团结合。相关生物研究说明人体内部的DNA甲基化与多种疾病、人体健康状况(比如BMI、年龄、吸烟等)具有联系。表观基因组关联分析即是通过DNA甲基化数据探测与某种性状相关联的CpG位点。但是,DNA甲基化是具有细胞类型特异性的,也就是说对于某个CpG位点,它在不同细胞类型中的甲基化程度会有所不同,从而可能只在某些特定细胞类型下才和性状有关联。HIRE的提出对探测出这样具有细胞类型特异性的关联CpG位点提供了可能。
在表观基因组中,一个重要的基本概念是DNA甲基化,它是指DNA序列上CpG二核苷酸的胞嘧啶与甲基基团结合。相关生物研究说明人体内部的DNA甲基化与多种疾病、人体健康状况(比如BMI、年龄、吸烟等)具有联系。表观基因组关联分析即是通过DNA甲基化数据探测与某种性状相关联的CpG位点。但是,DNA甲基化是具有细胞类型特异性的,也就是说对于某个CpG位点,它在不同细胞类型中的甲基化程度会有所不同,从而可能只在某些特定细胞类型下才和性状有关联。HIRE的提出对探测出这样具有细胞类型特异性的关联CpG位点提供了可能。

